Search

毛细管电泳(CE)技术可为靶向基因组分析提供精准明确的结果,极大程度帮助您降低试错成本。
利用毛细管电泳(CE)进行核酸分析主要通过两种方法实现:Sanger测序与片段分析。这两种技术均提供出从样本到结果的简易工作流程,既能实现高精度序列检测,又能保障快速获得分析报告。

Sanger测序
以单碱基分辨率实现精准测序判读的金标准技术,是靶向测序与验证性分析的理想选择。
了解Sanger测序如何支持多种应用场景:
下载Sanger测序指南 ›
探索简单易用的Sanger测序工作流程 ›
Applied Biosystems毛细管电泳产品为Sanger测序及片段分析提供端到端整体解决方案,涵盖基因分析仪器、试剂与软件三大核心组成。
InnoviGene Suite)是一套基于浏览器的一体化Sanger测序应用软件。它为您的数据分析提供了一套和谐组合的工具,以实现测序自动化和优化。


临床研究环境中的应用
Sanger测序与片段分析是从临床研究到临床参考实验室等多种临床环境中的基础性应用技术。
mRNA生产质控
在mRNA疗法及疫苗生产过程中,序列验证至关重要。选择Sanger测序技术,确保万无一失。
基因编辑验证
采用简易的Sanger测序与片段分析检测方案,实现对CRISPR、TALENs及先导编辑基因组工程的基因型验证。

细胞株鉴定
短串联重复序列(STR)基因分型技术,是验证人类细胞株真实性、评估储存人体组织与体液质量的关键工具。
其他毛细管电泳应用亮点
CRISPR-Cas9介导的基因组编辑技术正迅速普及至大多数生物科研工作者,即将为生物学及医疗健康领域带来革命性变革。赛默飞提供基因组编辑项目全流程解决方案。作为核心环节,SeqStudio基因分析仪专为Sanger测序分析设计,完美适配基因组编辑工作流程。其生成数据可直接兼容TIDE(插入缺失追踪分解)软件——该工具被广泛应用于基因组编辑效率分析。
通过HEK293细胞全细胞裂解液验证SeqStudio基因分析仪的实用性:在HPRT或relA基因座靶点区域引入随机缺失编辑后,将Sanger测序图谱上传至云平台,使用Sanger变异分析模块精确定位编辑位点(图1)。值得注意的是,断裂点下游出现的密集混合碱基峰可清晰指示编辑位置。利用TIDE软件分析这些图谱文件,测得该混合原代培养体系的编辑效率。正反向测序数据显示,各基因座缺失谱及发生频率高度一致(图2),此结果与英杰公司TOPO克隆联合Sanger测序的实验结论相互印证。
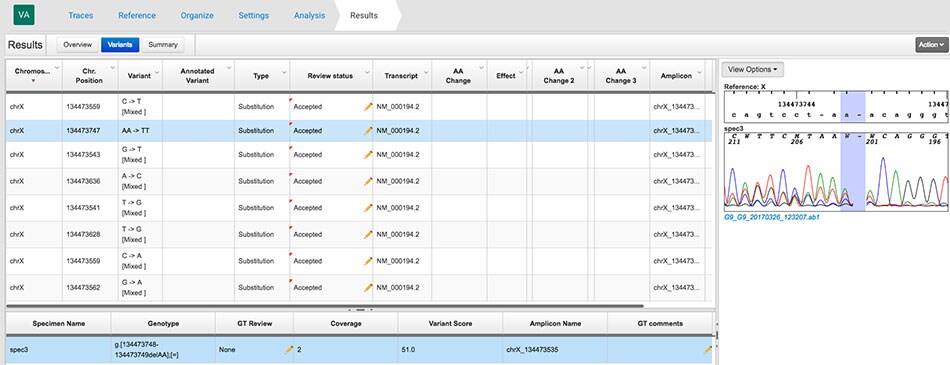
图1. 使用SeqStudio基因分析仪分析基因组编辑样本。通过云端Sanger变异分析应用检测人类HPRT基因座发生编辑的细胞混合群体。该应用不仅能识别正反链共有单核苷酸变异,还可精准定位基因组断裂事件(如本案例所示:断裂点下游出现混合序列群)。
A
B

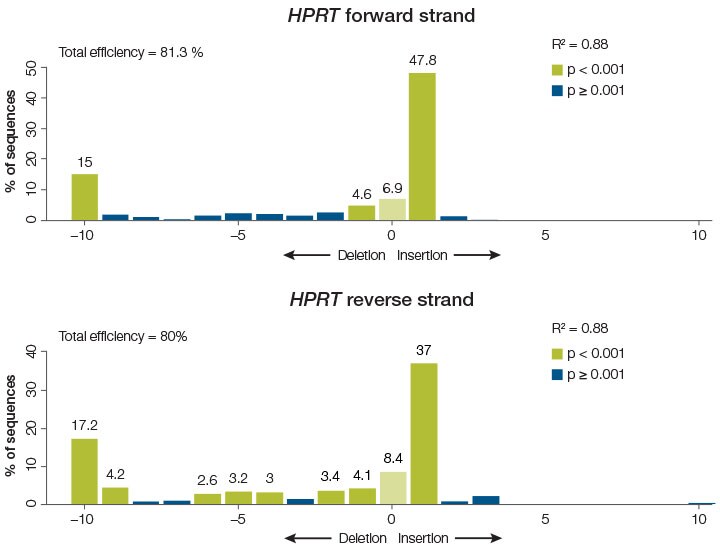
图2. TIDE软件分析SeqStudio生成的双基因座编辑数据。柱状图显示群体中特定核苷酸缺失/插入的比例。(A)HPRT基因座总体编辑效率约80%,(B)relA基因座总体编辑效率约20%。
在高等真核生物中,CpG二核苷酸基序的胞嘧啶5号碳位(5-meC)甲基化状态是细胞发育过程的关键表观遗传调控机制。因此,解析癌症等病变中DNA甲基化模式的变化具有重大研究价值,目前已发现众多基因组区域及特定位点的CpG甲基化状态直接影响细胞与组织生理功能。然而,利用福尔马林固定石蜡包埋(FFPE)样本开展回顾性研究时,样本核酸的降解状态构成主要技术障碍。开发适配FFPE组织的DNA甲基化分析方法,将为这类珍贵回顾性样本资源开辟全新研究路径。
25年来,Sanger测序始终是5-meC检测的金标准技术,可在长达400核苷酸的基因组DNA(gDNA)区域内实现多位点甲基化检测。当前最主流的5-meC检测方法是对gDNA进行亚硫酸氢盐处理。甲基化测序直接法(Methyl-Seq Direct)作为创新性亚硫酸盐测序方案,采用Applied Biosystems BigDye直接测序试剂盒与BigDye XTerminator纯化系统,完整工作流程(含亚硫酸盐转化、PCR扩增及DNA测序)仅需约7小时,其中手动操作时间约1小时。该方案完美兼容FFPE样本,使得利用存档样本开展肿瘤等病变回顾性研究成为可能。
为验证FFPE组织DNA分析可行性,研究者处理了两组癌变/正常组织配对样本。图1清晰展示了正常与肿瘤组织间甲基化模式的显著差异。
随着临床研究者日益通过液体活检收集并分析生物标志物,我们的第二篇应用指南阐述了一种基于Sanger测序的液体活检DNA甲基化分析方法。本研究通过选取更小的扩增子,实现对液体活检样本中高度片段化DNA的甲基化状态(阳性/阴性)检测。
应用指南:采用Sanger测序技术进行液体活检中的甲基化DNA分析
本系列指南通过赛默飞世尔提供的试剂、仪器及软件,验证了直接PCR测序工作流程在液体活检、细胞系、FFPE(福尔马林固定石蜡包埋)组织这些样本类型中的可行性,该流程适用于对经亚硫酸盐处理的DNA进行甲基化分析。
人类疾病机理研究高度依赖于体外培养的传代细胞株分析。然而,细胞培养过程中频发的细胞系错误鉴定或交叉污染问题日益受到关注。通过分析高度变异的短串联重复序列(STR)这一特异性遗传"指纹",可精准验证细胞株身份。SeqStudio平台与赛默飞细胞株鉴定解决方案深度整合:Applied Biosystems Identifiler Plus试剂盒适用于纯化DNA样本,Identifiler Direct试剂盒则支持粗提DNA样本,二者均能分析16个常用的人类STR基因座以验证细胞株真实性。配套的GeneMapper分析软件兼容SeqStudio仪器数据,可通过ATCC等STR数据库进行结果比对认证。
案例研究:《应用Identifiler STR分型试剂盒验证iPSCs与供体匹配性》
应用指南:《基于Identifiler试剂盒与毛细管电泳平台的人类细胞株鉴定》
网络研讨会:《人类细胞株全流程鉴定解决方案》
为证实SeqStudio基因分析仪在细胞株鉴定工作流中的实用性,对五种常用人类细胞株进行STR等位基因分析,仅需300 pg基因组DNA(gDNA)即可完成身份验证。在M4A4GFP细胞群中掺入梯度浓度HeLa细胞,采用Identifiler Direct试剂盒检测。实验证明:当HeLa细胞占比低至10%时,其特异性等位基因仍可被检出(图1)。因此,结合Identifiler系列试剂盒,SeqStudio可成为细胞株鉴定解决方案的核心设备。
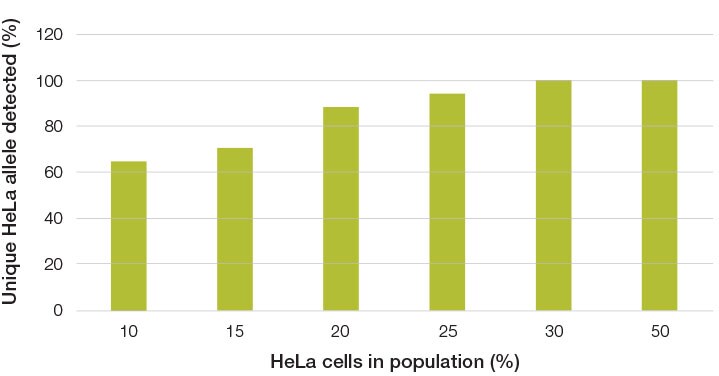
图1. 使用SeqStudio基因分析仪进行细胞污染分析。将HeLa与M4A4GFP细胞悬液(浓度5×10⁵个细胞/mL)按指定比例混合后点样于核酸采集卡。当污染细胞占比约20%时,SeqStudio基因分析仪可实现高置信度检测;部分HeLa特异性等位基因在10%占比时仍可识别。
临床研究者可采用SeqStudio基因分析仪,以金标准品质检测并验证肿瘤组织中的突变等位基因。该系统通过以下工具简化Sanger测序流程:
- 预装运行模块:专为福尔马林固定石蜡包埋(FFPE)组织提取的片段化DNA优化
- 云端NGC模块:基于Sanger测序图谱快速验证二代测序(NGS).vcf文件中的变异位点
- 配套Minor Variant Finder(MVF)软件:可检测低至5%频率的等位基因变异
- BigDye直接测序/BigDye XTerminator纯化试剂:实现"单管完成"测序与纯化
应用指南:《基于Sanger测序的FFPE样本低丰度体细胞突变检测》
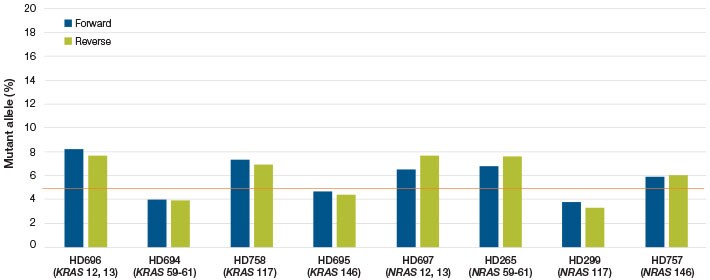
通过10例FFPE肿瘤样本的gDNA分析(覆盖4个热点区域),评估SeqStudio基因分析仪的突变检出能力。采用Ion Torrent Oncomine肿瘤靶向检测组合进行NGS分析,使用BigDye直接测序试剂/MVF软件进行Sanger测序。数据显示在9%-70%等位基因频率范围内,两种方法检测结果高度一致(图1)
图1. SeqStudio仪器对FFPE样本的突变频率分析
Ion Torrent测序与使用SeqStudio基因分析仪Sanger测序在十组肿瘤样本、四个热点区域的突变频率相关性分析。
采用预置Sanger测序引物的96孔板(靶向KRAS/NRAS常见致癌突变),SeqStudio基因分析仪在1 ng FFPE来源DNA中精准测定等位基因频率(图2A)。该结果证实:即使面对稀有等位基因检测,SeqStudio仍能确保FFPE组织分析的准确性。
NGC云平台通过按扩增子/样本归类Sanger测序图谱,并将其与.vcf文件的候选变异序列精准定位,极大简化NGS变异验证流程。图2B示例中,我们通过Sanger测序成功验证Oncomine检测出的NRAS突变(p.丙氨酸59苏氨酸),证实NGC应用在肿瘤研究流程中的实用性。
A
B
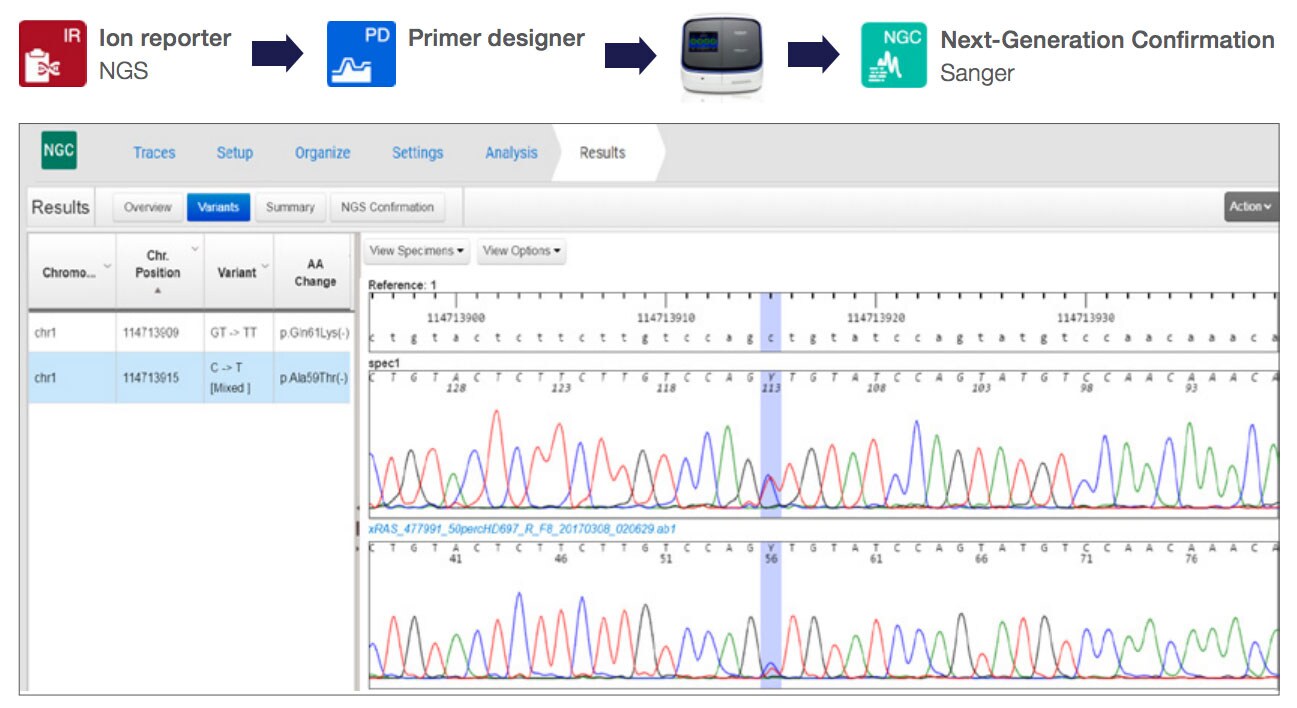
图2. SeqStudio与NGC应用的变异分析/验证功能。(A) 将8例含RAS热点突变的FFPE样本稀释至5%等位基因频率,经96孔板引物检测显示:所有突变频率测量值准确(黄色基线为5%,10%与50%稀释组结果类似);(B) NGS变异验证流程:基于Ion Reporter软件生成的.vcf文件,通过Primer Designer定制目标位点引物,经SeqStudio测序后由NGC云平台自动标注.vcf文件与Sanger图谱的共有变异。
基因组数据的便捷获取为未知样本的物种鉴定开辟了新途径——通过对"指纹"基因座进行DNA测序实现。例如,Applied Biosystems MicroSEQ系列试剂盒通过Sanger测序核糖体DNA(rDNA)序列,显著简化了原核生物与真菌的鉴定流程。同理,真核生物可利用线粒体特异基因座作为标识位点,该策略已在"生命条形码计划"(barcodeoflife.org)中成功应用,为快速鉴定未知真核样本提供有效手段。
采用ATCC标准微生物基因组DNA样本,配合Applied Biosystems MicroSEQ 500 PCR试剂盒与SeqStudio基因分析仪进行测序,将所得序列与BLAST数据库比对:所有测序反应均以最高置信度准确鉴定微生物物种;使用鱼类线粒体CO1基因引物检测鱼类样本,BLAST最佳匹配结果与物种完全一致(表1数据证实SeqStudio平台的物种鉴定可靠性)。
| Number of organisms | Number of queries | Percent correct | |
|---|---|---|---|
| Microoranisms | 24 | 48 | 100 |
| Piscine organisms | 12 | 24 | 100 |
表1. SeqStudio基因分析仪物种鉴定准确性。检测方法:微生物采用16S rDNA引物+MicroSEQ试剂盒(BigDye末端试剂v1.1);鱼类采用线粒体CO1引物+BigDye末端试剂v3.1。
应用指南:《16S直接检测法:基于SeqStudio基因分析仪的16S基因测序与MicrobeBridge软件微生物鉴定》
基因组不稳定性是多种癌症的标志性特征,常源于DNA错配修复(MMR)酶功能失调。由于至少涉及11个MMR相关基因座,全面检测所有位点的功能失活事件过程复杂、耗时且昂贵。此外,基于免疫组化(IHC)的MMR分析仅能实现半定量,其主观判读结果存在个体差异。因此,检测MMR失调的功能性后果——微卫星不稳定性(MSI)更具临床价值,可通过PCR联合片段分析的分子检测方案实现。
TrueMark MSI检测试剂盒采用扩展型13位点微卫星标志物组合,并包含2个高变短串联重复序列(STR)用于样本溯源。通过5个荧光通道分析13个基因座,显著提升MSI状态判定的序列多样性。该方案已针对SeqStudio及3500系列基因分析仪优化,配套TrueMark MSI分析软件无需癌旁正常组织对照即可实现自动化判读。
采用317例商业来源的FFPE肿瘤样本(结直肠癌、胃癌、子宫内膜癌等),利用SeqStudio和3500xl基因分析仪进行TrueMark MSI检测:成功将肿瘤分为稳定型(MSS)、低度不稳定型(MSI-L)和高度不稳定型(MSI-H)(表1);结直肠癌/胃癌/子宫内膜癌的MSI-H检出率与文献报道一致;在乳腺癌、腹腔肿瘤及肺癌等其他癌种中成功检出MSI,证实方案的广泛适用性。
| Tumor type | Number of samples | MSS | MSI-L | MSI-H |
|---|---|---|---|---|
| Colon | 73 | 47 (64%) | 0 | 26 (36%) |
| Gastric | 50 | 41 (82%) | 2 (4%) | 7 (14%) |
| Endometrial | 173 | 136 (79%) | 14 (8.1%) | 23 (12%) |
| Other | 21 | 17 (81%) | 1 (5%) | 3 (14%) |
表1. 多癌种MSI状态分析。检测标准:无基因座不稳定=MSS;1-4个基因座不稳定=MSI-L;≥5个基因座不稳定=MSI-H
拷贝数变异引发的遗传性疾病研究中,多重连接依赖性探针扩增(MLPA)是广泛应用的技术。该技术由MRC Holland公司开发并商业化,可同时分析多达50对靶向相邻基因座的探针。SeqStudio基因分析仪凭借高动态范围、精密的片段大小分析及峰高保真度,成为MLPA探针扩增产物分析的理想平台,其检测结果兼容MRC Holland Coffalyzer.Net数据分析软件。
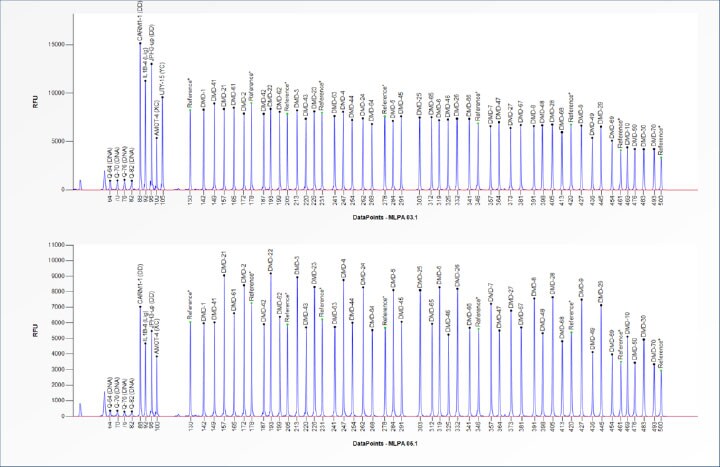
采用MRC Holland P034 DMD试剂盒,通过SeqStudio基因分析仪分析已知携带Duchenne型肌营养不良(DMD)基因2-30号外显子重复的DNA样本:通过峰高与相对大小数据精准定位重复区域(图1);大小片段缺失探针检测结果同样精准;证明SeqStudio可作为CNV区域MLPA研究的集成化工具。
A
B
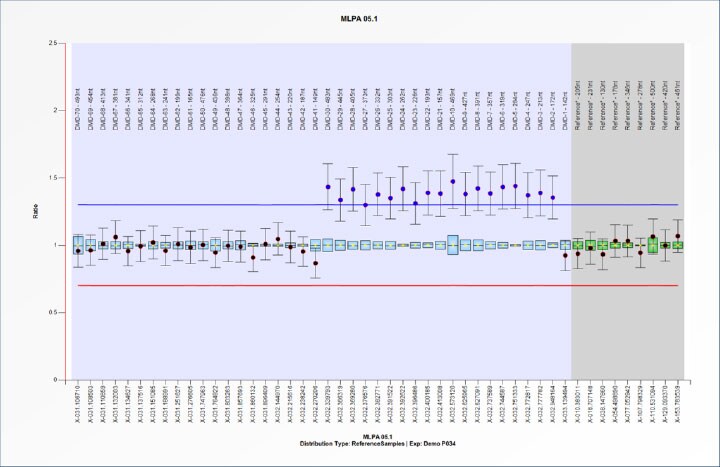
图1. SeqStudio基因分析仪MLPA法CNV分析。(A) DMD基因CNV检测片段图谱:下方样本(患者)部分片段峰高显著高于上方(正常对照),提示该区域DNA过量;(B) DMD基因座探针比值图:2-30号外显子探针比值显著升高(跨越蓝色阈值线提示拷贝数由2增至3)
Sanger测序最核心的应用之一是对亚克隆至质粒载体的插入片段进行分析。Applied Biosystems BigDye末端终止法作为Sanger测序的行业金标准,已成为质粒测序工作流程的关键环节。SeqStudio基因分析仪多项创新特性为常规质粒测序提供显著优势:
- 预置测序模块:针对短读长(<300 bp)、中读长(500 bp)及长读长(>600 bp)优化
- 可自定义方案:支持仪器端个性化参数设置
- 专属卡夹管理:可更换卡夹适配不同项目及用户需求
- 云端质控应用:提供直观的测序图谱分析工具
- 远程协作功能:支持云端监控、访问及数据共享
采用M13引物与BigDye末端终止试剂v3.1对pGEM7zf+质粒进行测序,通过赛默飞云平台Sanger质控模块分析结果(图1):16孔板样本经4次独立进样分析;各样本图谱质量评分、峰下峰(PUP)值、连续读长(CRL)及高质量读长(QV20+)指标高度一致;反向链测序及v1.1试剂验证结果同样优异。证明SeqStudio基因分析仪可产出超高精度质粒测序数据。
A

B
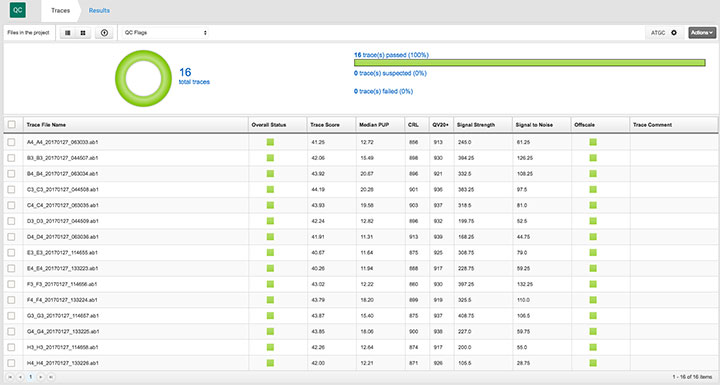
图1. Sanger质控云应用分析结果。(A) SeqStudio仪器运行完成后实时显示序列文件及碱基质量评分;(B) 16组pGEM7zf+测序反应结果云端分析:关键质量参数在全部样本间高度一致。*注:CRL=连续读长;QV20+=质量值>20的核苷酸数量*
指由多核苷酸重复序列构成的DNA突变类型。这类高GC含量区域通常难以扩增与表征,短串联重复序列(STRs)即属此类——包含1-6个核苷酸的长片段重复,与30余种遗传性疾病密切相关。
毛细管电泳(CE)片段分析技术凭借独特优势成为疾病相关DNA重复区域检测的理想方案。针对高GC含量区域的分析难点,本研究验证了SeqStudio基因分析仪配合Asuragen AmplideX™ PCR/CE FMR1/C9orf72试剂盒的进样与运行参数,成功实现:
- FMR1基因:扩增异常导致脆性X染色体综合征
- C9orf72基因:扩增引发额颞叶痴呆或肌萎缩侧索硬化(ALS)
技术白皮书:《SeqStudio基因分析仪联合AmplideX试剂实现三核苷酸重复扩增高效分析》
AmplideX试剂采用重复引物PCR与毛细管电泳联用技术,全长扩增子覆盖STR区域,重复引物产物提供致病等位基因验证性峰图。该方案在Applied Biosystems 3130/3500/3730平台已获75+文献验证,而SeqStudio仪器的短阵列与POP-1胶聚合物需优化参数以实现同等性能。
在SeqStudio系统上,片段分析模块(FragAnalysis) 与长片段分析模块(LongFragAnalysis) 对信号强度的影响相当。然而,使用片段分析模块虽获得更高信号强度(约提升50%),但出现信号过饱和的目的基因峰分裂现象。鉴于FMR1和C9orf72基因峰在两种模块中均存在此共性,最终选用长片段分析模块作为基础方案,参数设置为:进样时间:2秒;进样/运行电压:6 kV;运行时间:3,300秒(55分钟)。该条件产生的谱图与3500xL仪器高度一致,后续精密度与灵敏度分析均基于此配置(图1)。
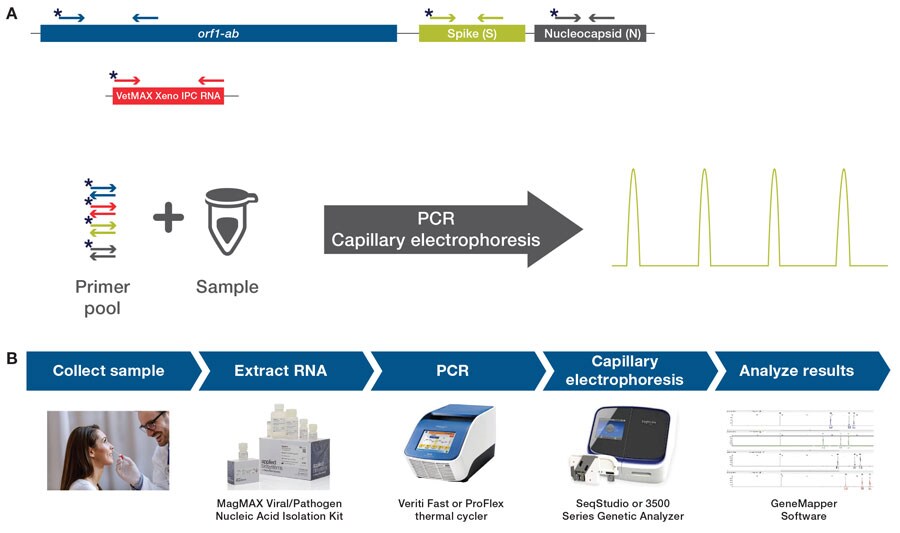
传统多重qPCR方案因检测通量有限,在需筛查大量靶标或病原体时存在瓶颈。基于毛细管电泳(CE)的片段分析技术可单次检测样本中多种综合征相关病原体。
我们开发了一种创新工作流程(图1),通过片段分析法同步检测病原体病毒的多重扩增子:靶标设计:以SARS-CoV-2为例,采用合成DNA阳性对照界定目标扩增子片段大小;灵敏度验证:使用梯度浓度SARS-CoV-2 RNA基因组验证方法灵敏度。
应用指南:《病毒病原体(含SARS-CoV-2)多重靶标片段分析检测方案》
实验方案:《基于片段分析的SARS-CoV-2 RNA检测》
了解更多:SARS-CoV-2研究的Sanger测序与片段分析解决方案
简而言之,在此应用中,确定合适的靶标序列后,扩增子应设计为150-500个核苷酸长度。每个扩增子需具有独特长度,与其他扩增子至少相差5个核苷酸。正向引物应在5´端标记荧光染料,反向引物则不标记。随后使用单管式系统(例如TaqMan Fast Virus 1-Step Master Mix)建立逆转录聚合酶链反应(RT-PCRs),该系统允许在同一反应管内完成cDNA合成及终点PCR。核酸样本经PCR扩增后,通过毛细管电泳(CE)分离所得片段。预期大小的病原体特异性片段出现与否决定样本中病原体的存在状态(表1),峰高可作为样本中靶标丰度的半定量指标。
| 表1. SARS-CoV-2序列片段分析结果峰图判读建议。类似判定标准已用于基于qPCR的新冠病毒检测,其他检测方案的参数可根据具体检测需求及目标病原体特征进行设定。 | ||
| Xeno RNA control peak | SARS-CoV-2 RNA peaks | Interpretation results |
| Present | None | Negative—no SARS CoV-2 RNA detected |
| Present | 3 peaks | Positive—SARS CoV-2 RNA detected |
| Present | 2 peaks | Positive—SARS CoV-2 RNA detected |
| Present | 1 peak | Indeterminate—retest the same purified sample |
| No peak | No peak | Invalid—no SARS-CoV-2 RNA detected; retest the same purified sample |
检测单核苷酸多态性(SNP)的能力对于解析基因组如何影响生物表型至关重要。Applied Biosystems SNaPshot多重检测系统专为SNP变异分析而开发:通过片段分析技术检测与特定等位基因对应的、可定制化且颜色编码的不同长度片段。SeqStudio系统新增功能显著优化SNaPshot分析流程,包括:内置报告功能:自动生成片段长度与峰面积分析结果;混合检测模式:支持同板进行片段分析与测序反应,单次运行即可完成SNP分型与Sanger测序。
为了应用验证,收集FFPE保存的肿瘤切片gDNA,采用SNaPshot多重试剂盒(靶向KRAS G12X/G13X等位基因探针)进行分析:SeqStudio仪器清晰呈现不同等位基因的存在状态与精准判读(图1)。技术提示:因聚合物化学性质差异,所有峰迁移值较其他平台略有偏移。为确保等位基因无歧义匹配,大规模分析前需进行已知等位基因校准。
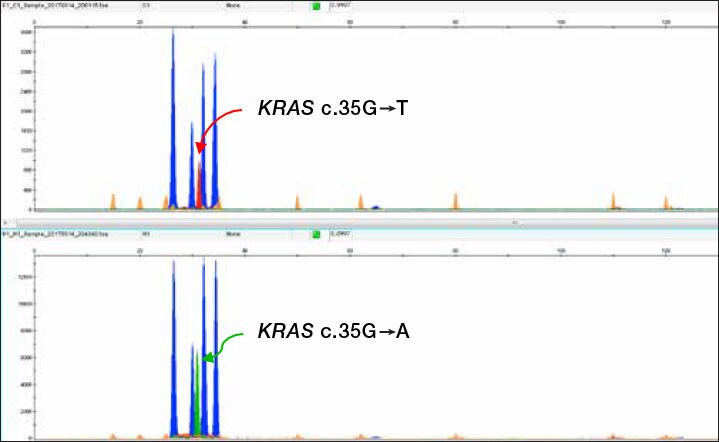
图1. SeqStudio平台SNaPshot试剂盒SNP分析。两例FFPE来源肿瘤DNA(各1纳克)经SNaPshot多重试剂盒(KRAS特异性引物)处理后片段分析结果:蓝色峰:KRAS c.34/c.35/c.37/c.38位点野生型等位基因;红色峰(上图):样本存在KRAS c.35G>T突变‘’绿色峰(下图):同一位点的其他等位基因变异。
Applied Biosystems SeqStudio基因分析仪(HID版)提供金标准STR片段分析与Sanger测序的一键式操作。该仪器广泛兼容各类法医STR试剂盒与应用场景,支持多用户协作。延续Applied Biosystems仪器卓越的数据质量与服务支持,助您获取值得信赖的精准结果。
法医遗传学应用方向:SeqStudio基因分析仪在研究场景中的人类身份鉴定
毛细管电泳(CE)与二代测序(NGS)技术在法医DNA分析中的联合应用
聚焦科学家
聚焦科学家
Athena Starlard-Davenport, PhD 纳西大学健康科学中心(UTHSC)
主题:非裔美国女性间乳腺癌的差异:Sanger测序提供的见解
视频:看Sanger测序和片段分析如何超越您的期待!
更多资源
支持
For Research Use Only. Not for use in diagnostic procedures.







